
Post-doctorante
Laboratoire PRISM - Inserm U1192
Villeneuve d´Ascq
Après plus de dix heures de voyage en voiture, train et avion, nous sommes arrivés à Dresden, Allemagne. Une fois arrivé, nous nous sommes tranquillement installés à l’hôtel, où nous avons retrouvé plusieurs participants du congrès. Étant donné la taille modeste de la ville, les participants à HUPO se reconnaissent facilement.
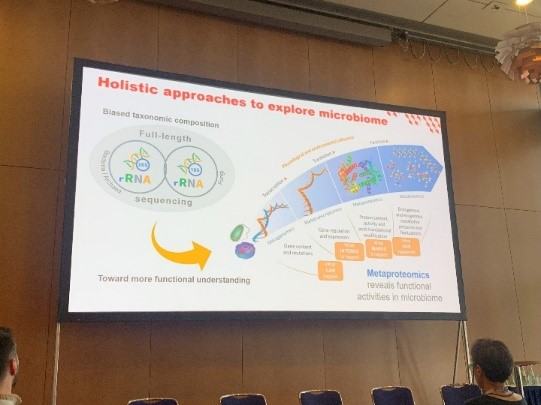 Le congrès nous a tenus bien occupés du dimanche 20 au jeudi 24 octobre. J’ai commencé le premier jour avec le Pre-congress Workshop: Delving into biodiversity’s depths: integrating new model organisms and unravelling the mysteries of unknown proteins. Cette session a débuté avec la présentation de Jean Armengaud intitulée Holobiont models: improving our understanding of biological complexity, où il a abordé l’étude du microbiome dans différents organismes et la vision globale des organismes. Ensuite, Tristan Cardon, membre de notre laboratoire (PRISM U1192), a présenté ses travaux sur les protéines alternatives, également appelées « Ghost Proteins », à travers son intervention intitulée Ghost proteins: an analogy to novel model organisms for deciphering protein functions.
Le congrès nous a tenus bien occupés du dimanche 20 au jeudi 24 octobre. J’ai commencé le premier jour avec le Pre-congress Workshop: Delving into biodiversity’s depths: integrating new model organisms and unravelling the mysteries of unknown proteins. Cette session a débuté avec la présentation de Jean Armengaud intitulée Holobiont models: improving our understanding of biological complexity, où il a abordé l’étude du microbiome dans différents organismes et la vision globale des organismes. Ensuite, Tristan Cardon, membre de notre laboratoire (PRISM U1192), a présenté ses travaux sur les protéines alternatives, également appelées « Ghost Proteins », à travers son intervention intitulée Ghost proteins: an analogy to novel model organisms for deciphering protein functions.  Cette nouvelle vision du protéome, avec l'identification de protéines alternatives dans différents types de cancer représente une mine d'informations inexplorées à partir de laquelle de nouveaux biomarqueurs pourraient émerger. De plus, le lendemain, lors de la session des Poster Presentations of HUPO & EuPA initiatives, un poster a été présenté sur l’Initiative for Model Organism Proteomics (iMOP), à laquelle notre laboratoire contribue à travers la participation de Tristan Cardon et Jean Armengaud, aux côtés d’autres scientifiques (Fabrice Bertile, Sabine Matallana-Surget, Andreas Tholey et Susana Cristobal). L’initiative iMOP, portée par HUPO et EuPA, vise à soutenir l’étude des protéines essentielles à la santé humaine et à l’environnement en utilisant des modèles biologiques innovants et des approches avancées comme la protéomique comparative et la protéogénomique.
Cette nouvelle vision du protéome, avec l'identification de protéines alternatives dans différents types de cancer représente une mine d'informations inexplorées à partir de laquelle de nouveaux biomarqueurs pourraient émerger. De plus, le lendemain, lors de la session des Poster Presentations of HUPO & EuPA initiatives, un poster a été présenté sur l’Initiative for Model Organism Proteomics (iMOP), à laquelle notre laboratoire contribue à travers la participation de Tristan Cardon et Jean Armengaud, aux côtés d’autres scientifiques (Fabrice Bertile, Sabine Matallana-Surget, Andreas Tholey et Susana Cristobal). L’initiative iMOP, portée par HUPO et EuPA, vise à soutenir l’étude des protéines essentielles à la santé humaine et à l’environnement en utilisant des modèles biologiques innovants et des approches avancées comme la protéomique comparative et la protéogénomique.
 De plus, Tristan n’était pas le seul à représenter le laboratoire, un autre membre de l’équipe, Léa Ledoux, a été invitée à présenter ses travaux de recherche le lundi 21, lors de la session Spatial and Imaging Proteomics. Elle a exposé l’une des dernières technologies développées au laboratoire, le Backside, à travers une présentation intitulée Backside laser irradiation on histological slide sections for multi-omics applications: spot-by-spot LC-MS imaging. Devant une salle comble, Léa a détaillé une étude utilisant SpiderMass pour analyser simultanément protéines, lipides et métabolites avec une résolution de 220 µm. Cette approche permet une cartographie 2D précise et l’identification de plus de 5000 protéines. Optimisée pour divers tissus et lames histologiques, cette technologie simplifie les workflows analytiques tout en ouvrant de nouvelles perspectives sur l’étude de l’hétérogénéité tissulaire.
De plus, Tristan n’était pas le seul à représenter le laboratoire, un autre membre de l’équipe, Léa Ledoux, a été invitée à présenter ses travaux de recherche le lundi 21, lors de la session Spatial and Imaging Proteomics. Elle a exposé l’une des dernières technologies développées au laboratoire, le Backside, à travers une présentation intitulée Backside laser irradiation on histological slide sections for multi-omics applications: spot-by-spot LC-MS imaging. Devant une salle comble, Léa a détaillé une étude utilisant SpiderMass pour analyser simultanément protéines, lipides et métabolites avec une résolution de 220 µm. Cette approche permet une cartographie 2D précise et l’identification de plus de 5000 protéines. Optimisée pour divers tissus et lames histologiques, cette technologie simplifie les workflows analytiques tout en ouvrant de nouvelles perspectives sur l’étude de l’hétérogénéité tissulaire.
 Dans cette même session, une autre présentation particulièrement intéressante était celle Frederik Post : Deep visual proteomics advances human colon organoid models by revealing a switch to an in vivo-like phenotype upon xenotransplantation. Cette étude améliore la technologie Deep visual proteomics pour cartographier le protéome de la muqueuse colique humaine. Les résultats montrent que les organoïdes humains transplantés dans des colons murins retrouvent un phénotype semblable aux cellules épithéliales intestinales in vivo, offrant des perspectives pour le traitement des maladies inflammatoires de l'intestin. Cette thématique me parle particulièrement, car je travaille moi-même sur des organoïdes de cancer du sein.
Dans cette même session, une autre présentation particulièrement intéressante était celle Frederik Post : Deep visual proteomics advances human colon organoid models by revealing a switch to an in vivo-like phenotype upon xenotransplantation. Cette étude améliore la technologie Deep visual proteomics pour cartographier le protéome de la muqueuse colique humaine. Les résultats montrent que les organoïdes humains transplantés dans des colons murins retrouvent un phénotype semblable aux cellules épithéliales intestinales in vivo, offrant des perspectives pour le traitement des maladies inflammatoires de l'intestin. Cette thématique me parle particulièrement, car je travaille moi-même sur des organoïdes de cancer du sein.
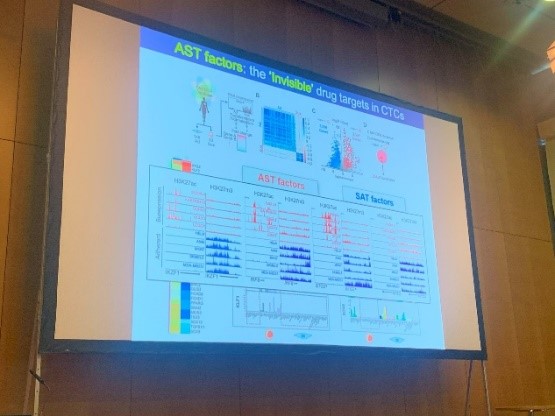 En réalité, le congrès a été rempli d'incroyables présentations, et bien que je ne puisse pas toutes les citer, quelques-unes m'ont particulièrement intéressée. Pour en citer quelques-unes, lors de la session Multi-omics approaches and data integration, Hyun Woo Park a présenté Multi-omics analysis in CTC-based liquid biopsy, montrant l'étude de l'AST (Adherent-to-Suspension Transition), qui reprogramme les cellules tumorales en cellules tumorales circulantes (CTC) lors de la métastase. L'AST, induite par des régulateurs transcriptionnels hématopoïétiques, permet la dissociation cellule-matrice, la résistance à l'anoïkose et l'évasion immunitaire. Cibler les facteurs de l'AST pourrait réduire la formation de CTC et la métastase, offrant ainsi des thérapies anti-métastatiques potentielles.
En réalité, le congrès a été rempli d'incroyables présentations, et bien que je ne puisse pas toutes les citer, quelques-unes m'ont particulièrement intéressée. Pour en citer quelques-unes, lors de la session Multi-omics approaches and data integration, Hyun Woo Park a présenté Multi-omics analysis in CTC-based liquid biopsy, montrant l'étude de l'AST (Adherent-to-Suspension Transition), qui reprogramme les cellules tumorales en cellules tumorales circulantes (CTC) lors de la métastase. L'AST, induite par des régulateurs transcriptionnels hématopoïétiques, permet la dissociation cellule-matrice, la résistance à l'anoïkose et l'évasion immunitaire. Cibler les facteurs de l'AST pourrait réduire la formation de CTC et la métastase, offrant ainsi des thérapies anti-métastatiques potentielles.
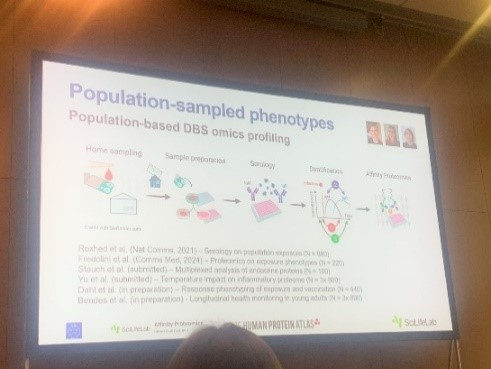 Lors de la session Population Proteomics in Health and Disease, Annika Bendes a présenté l'étude Trans-pandemic population profiling in young adults. Cette étude a collecté des échantillons de sang capillaire en envoyant des kits de prélèvement à domicile à 2000 habitants sélectionnés au hasard (âgés de 20 à 74 ans) de la région métropolitaine de Stockholm en avril 2020. Entre 2020 et 2022, environ 800 participants de la cohorte suédoise BAMSE ont fourni des échantillons de sang séché (DBS) analysés par sérologie multi-analytes et protéomique pour mesurer la réponse immunitaire contre 38 antigènes du SARS-CoV-2 et 365 protéines liées à l'inflammation. Les résultats ont montré des réponses immunitaires attendues à l'infection et à la vaccination, avec des signatures protéiques variables entre les individus, et ont souligné l'importance du délai entre le prélèvement et la vaccination sur l'intensité de la réponse. Cette étude montre la faisabilité d'utiliser les DBS pour analyser les réponses immunologiques et protéomiques aux vaccins et infections, offrant des informations sur l'influence des conditions préexistantes et acquises sur la santé.
Lors de la session Population Proteomics in Health and Disease, Annika Bendes a présenté l'étude Trans-pandemic population profiling in young adults. Cette étude a collecté des échantillons de sang capillaire en envoyant des kits de prélèvement à domicile à 2000 habitants sélectionnés au hasard (âgés de 20 à 74 ans) de la région métropolitaine de Stockholm en avril 2020. Entre 2020 et 2022, environ 800 participants de la cohorte suédoise BAMSE ont fourni des échantillons de sang séché (DBS) analysés par sérologie multi-analytes et protéomique pour mesurer la réponse immunitaire contre 38 antigènes du SARS-CoV-2 et 365 protéines liées à l'inflammation. Les résultats ont montré des réponses immunitaires attendues à l'infection et à la vaccination, avec des signatures protéiques variables entre les individus, et ont souligné l'importance du délai entre le prélèvement et la vaccination sur l'intensité de la réponse. Cette étude montre la faisabilité d'utiliser les DBS pour analyser les réponses immunologiques et protéomiques aux vaccins et infections, offrant des informations sur l'influence des conditions préexistantes et acquises sur la santé.
La dernière journée, nous avons assisté à des présentations très intéressantes sur les avancées en protéomique et leurs applications médicales, abordant des sujets tels que l'impact de l'alcool sur le microenvironnement du cancer buccal (Adriana Franco Paes Leme), l’utilisation de la protéomique à haut débit pour analyser les populations humaines et découvrir des médicaments (Markus Ralser), ainsi que des études cliniques sur les biomarqueurs de l'obésité (Aleksandra Dudzik) et le diagnostic du cancer (Arthur Viodé). Il y a également eu des présentations sur l'immunologie, notamment sur l’immunopeptidomique, un sujet passionnant en ce moment avec un mélange des développements technologiques pour leur détection et les résultats (Anthony Purcell, Nicola Ternette, Adrianna Seredynska, Ilja Shapiro). De plus, Chen Li a présenté la plateforme CAN-IMMUNE, qui utilise des données génétiques pour créer des bibliothèques de marqueurs et aider à identifier des cibles pour développer des traitements personnalisés contre le cancer. Je ne pourrais jamais résumer toutes les excellentes présentations du congrès, car chacune d'elles apportait une perspective unique avec des discussions riches et variées.
 D'autre part, j'ai énormément apprécié les sessions de "Mentoring", qui abordent des sujets plus personnels liés à la recherche. Celle que j'ai préférée cette année à HUPO était la présentation sur "Mental Health: Building resilience to manage work and job search stress". Les trois présentateurs, Jennifer Geddes-McAlister, Adriana Franco Paes Leme et Stuart Cordwell, ont partagé leur parcours et nous ont donné des conseils concrets sur la manière de gérer cet aspect de la recherche.
D'autre part, j'ai énormément apprécié les sessions de "Mentoring", qui abordent des sujets plus personnels liés à la recherche. Celle que j'ai préférée cette année à HUPO était la présentation sur "Mental Health: Building resilience to manage work and job search stress". Les trois présentateurs, Jennifer Geddes-McAlister, Adriana Franco Paes Leme et Stuart Cordwell, ont partagé leur parcours et nous ont donné des conseils concrets sur la manière de gérer cet aspect de la recherche.

 Les sessions poster ont, elles, été particulièrement riches en échanges. J’ai eu l’opportunité de présenter mon poster intitulé "Characterization of breast cancer tumoroids subclones by proteomics for personalized medicine". Mon travail vise à dépasser les limitations des modèles traditionnels en développant des tumoroïdes de cancer du sein à partir de tissus humains et canins. Nous avons exploré l’hétérogénéité intratumorale à l’aide de la protéomique et de l’imagerie par spectrométrie de masse, montrant que certains sous-clones réagissent différemment aux traitements. Ces résultats ouvrent la voie à des stratégies thérapeutiques plus ciblées pour prévenir les rechutes. Ce congrès m'a permis de présenter ces résultats et d'échanger sur leurs implications pour la médecine personnalisée.
Les sessions poster ont, elles, été particulièrement riches en échanges. J’ai eu l’opportunité de présenter mon poster intitulé "Characterization of breast cancer tumoroids subclones by proteomics for personalized medicine". Mon travail vise à dépasser les limitations des modèles traditionnels en développant des tumoroïdes de cancer du sein à partir de tissus humains et canins. Nous avons exploré l’hétérogénéité intratumorale à l’aide de la protéomique et de l’imagerie par spectrométrie de masse, montrant que certains sous-clones réagissent différemment aux traitements. Ces résultats ouvrent la voie à des stratégies thérapeutiques plus ciblées pour prévenir les rechutes. Ce congrès m'a permis de présenter ces résultats et d'échanger sur leurs implications pour la médecine personnalisée.
Et pour finir, même si le congrès était riche en recherche, il l'était aussi en rencontres. Le lundi soir, les jeunes chercheurs ont conclu la journée avec l’événement ECR organisé autour d'un apéritif pendant lequel nous avons dîné ensemble. Ensuite, nous avons participé à un exercice de groupe où nous avons eu l'occasion de mélanger nos expériences et de discuter avec des personnes de différents horizons. Cet exercice a été très enrichissant, car il m’a permis de dialoguer avec des chercheurs d’autres disciplines, et d’avoir un moment convivial pour rencontrer de nouvelles personnes.

 Le mardi, après une journée bien remplie de présentations, la soirée s'est terminée par le dîner de gala organisé dans la salle du congrès, où nous avons mangé un excellent repas. Puis, une soirée dansante a eu lieu dans la salle plénière, totalement transformée pour l'occasion, offrant une ambiance complètement différente de celle des matinées. La soirée a été un grand succès, grâce à un DJ passionné, qui a fait danser tout le monde jusqu’à la fin de la nuit.
Le mardi, après une journée bien remplie de présentations, la soirée s'est terminée par le dîner de gala organisé dans la salle du congrès, où nous avons mangé un excellent repas. Puis, une soirée dansante a eu lieu dans la salle plénière, totalement transformée pour l'occasion, offrant une ambiance complètement différente de celle des matinées. La soirée a été un grand succès, grâce à un DJ passionné, qui a fait danser tout le monde jusqu’à la fin de la nuit.
Le congrès s'est finalement clôturé sur l’annonce de la prochaine édition, qui se tiendra a Toronto, Canada. Ce fut un moment de rires, marqué par le lancer de t-shirts de HUPO avec un bâton de hockey.
Pour conclure, je remercie la FPS pour leur bourse, qui a rendu ma participation possible, ainsi que HUPO pour le complément de bourse. Grâce à ces soutiens, j'ai pu participer à HUPO 2024 et assister à des présentations toutes aussi intéressantes les unes que les autres. Ce congrès a été une occasion exceptionnelle d'apprendre, d'échanger des idées et de renforcer les liens avec des jeunes chercheurs que j'estime énormément. En espérant revoir tout le monde très bientôt, rendez-vous à HUPO 2025 à Toronto !

